Fundamentos de los Microbiomas
El microbioma es el conjunto completo de microorganismos (la flora intestinal), sus genes y su microambiente (el hábitat) en un área específica. Las bacterias comprenden aproximadamente el 98 % del microbioma.1,2
Los subconjuntos del microbioma incluyen el viroma (virus), el micobioma (hongos) y el arqueoma (arqueas; estos organismos se asemejan a las bacterias, aunque son un dominio distinto).3 Es un entorno dinámico con interacciones complejas y metabolismos interconectados.4
El viroma comprende virus que infectan las bacterias (bacteriófagos) dentro del microbioma y las células huésped en el medioambiente.5,6 Los bacteriófagos comprenden la mayoría del viroma.5,6 Hasta la fecha, la investigación sobre el viroma es limitada,6,7 pero se han identificado cambios en el viroma fecal en perros con enteropatía crónica o diarrea aguda.5,6 De igual manera, la investigación publicada sobre el micobioma en mascotas también es limitada en este momento.8
Los genes presentes en el microbioma intestinal superan ampliamente los genes del huésped. En humanos, se estima que los genes microbianos los superan por más de 100 veces.9
El análisis metagenómico del microbioma intestinal de gatitos mostró que la cantidad de genes únicos identificados fue aproximadamente 108 veces mayor que la cantidad de marcos de lectura abiertos identificados en el genoma del gato.10
Los filos bacterianos de mayor cantidad en el microbioma intestinal son Firmicutes, Bacteroides y Fusobacterium, seguidos de Proteobacteria y Actinobacteria.11
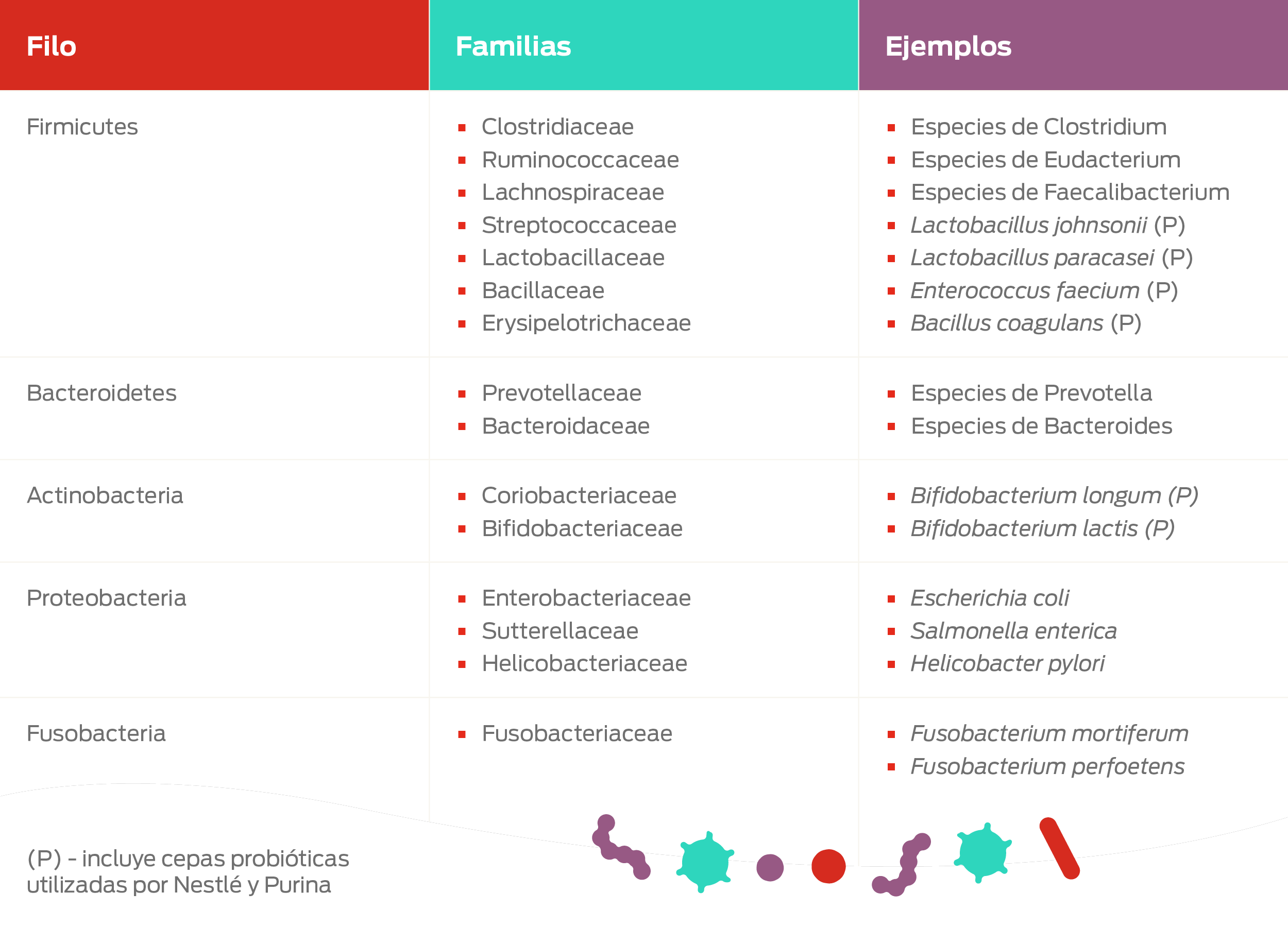
Proteobacteria es el filo más diverso en el microbioma intestinal e incluye varios patógenos oportunistas conocidos, como Escherichia coli, Klebsiella, Salmonella y Campylobacter, así como bacterias que cumplen funciones vitales en la homeostasis intestinal.12 El microbioma fecal felino puede ser más diverso que el del perro.12
Explorar Otras Áreas Del Foro De Microbiomas

Intervenciones Centradas en el Microbioma para las Afecciones de Salud de las Mascotas

Liderazgo de Nestlé y Purina en los Microbiomas
Obtén más información
- Barko, P.C., McMichael, M.A., Swanson, K.S., Williams, D.A. (2018). The gastrointestinal microbiome: a review. Journal of Veterinary Internal Medicine, 32, 9–25. doi: 10.1111/jvim.14875
- Marchesi, J. R. & Ravel, J. (2015). The vocabulary of microbiome research: a proposal. Microbiome, 3, 31. doi: 10.1186/s40168-015-0095-5
- Kim, J. Y., Whon, T. W., Lim, M. Y., Kim, Y. B., Kim, N., Kwo, M.-S.,…Na, Y.-D. (2020). The human gut archaeome: identification of diverse haloarchaea in Korean subjects. Microbiome, 8, 114. doi: 10.1186/s40168-020-00894-x
- Seth, E. C., & Taga, M. E. (2014). Nutrient cross-feeding in the microbial world. Frontiers in Microbiology, 5, 350. doi: 10.3389/fmicb.2014.00350
- Moreno, P. S., Wagner, J., Mansfield, C. S., Stevens, M., Gilkerson, J. R., & Kirkwood, C. D. (2017). Characterization of the canine faecal virome in healthy dogs and dogs with acute diarrhoea using shotgun metagenomics. PLoS ONE, 12(6), e0178433. doi: 10.1371/journal.pone.0178433
- Moreno, P. S., Wagner, J., Kirkwood, C. D., Gilkerson, J. R., & Mansfield, C. S. (2018). Characterization of the fecal virome in dogs with chronic enteropathy. Veterinary Microbiology, 221, 38–43. doi: 10.1016/j.vetmic.2018.05.020
- Zhang, W., Li, L., Deng, X., Kapusinszky, B., Pesavento, P. A., Delwart, E. (2014). Faecal virome of cats in an animal shelter. Journal of General Virology, 95, 2553–2564. doi: 10.1099/vir.0.069674-0
- Foster, M. L., Dowd, S. E., Stephenson, C., Steiner, J. M., & Suchodolski, J. S. (2013). Characterization of the fungal microbiome (mycobiome) in fecal samples from dogs. Veterinary Medicine International, 2013, 658373. doi: 10.1155/2013/658373
- Richards, P., Thornberry, N. A., & Pinto, S. (2021). The gut-brain axis: Identification of new therapeutic approaches for Type 2 diabetes, obesity, and related disorders. Molecular Metabolism, E pub ahead of print. doi: 10.1016/j.molmet.2021.101175
- Deusch, O., O’Flynn, C., Colyer, A., Morris, P., Allaway, D., Jones, P. G., & Swanson, K. S. (2014). Deep Illumina-based shotgun sequencing reveals dietary effects on the structure and function of the fecal microbiome of growing kittens. PLoS ONE, 9(7), e101021. doi: 10.1371/ journal.pone.0101021
- Suchodolski, J. S. (2011). The intestinal microbiota of dogs and cats: A bigger world than we thought. Veterinary Clinics of North America Small Animal Practice, 41, 261–272. doi: 10.1016/j.cvsm.2010.12.006
- Belas, A., Marques, C., & Pomba, C. (2020). The gut microbiome and antimicrobial resistance in companion animals. In Duarte, A. & Lopes da Costa, L. (Eds.), Advances in Animal Health, Medicine and Production (1st ed.), pp. 233–245. Springer International Publishing
