Notions de Base sur le Microbiome
Le microbiome désigne l'ensemble complet des micro-organismes (le microbiote), de leurs gènes et de leur microenvironnement (habitat) dans une zone spécifique. Les bactéries constituent environ 98 % du microbiome.1,2
Les sous-ensembles du microbiome sont le virome (les virus), le mycobiome (les champignons) et l'archéome (archées : organismes qui ressemblent à des bactéries, mais qui représentent un domaine distinct).3 C'est un environnement dynamique avec des interactions complexes et des métabolismes interconnectés.4
Le virome comprend des virus qui infectent les bactéries (bactériophages) du microbiome ainsi que les cellules de l'hôte dans l'environnement.5,6 Les bactériophages constituent la majorité du virome.5,6 À ce jour, la recherche sur le virome est limitée.6,7 Mais des changements dans le virome fécal ont été identifiés chez les chiens atteints d'entéropathie chronique ou de diarrhée aiguë.5,6 De même, le corpus des études publiées sur le mycobiome des animaux de compagnie est également limité à l'heure actuelle.8
Les gènes présents dans le microbiome intestinal dépassent de loin les gènes de l'hôte. Chez l'homme, on estime que les gènes microbiens sont plus de 100 fois plus nombreux.9
L'analyse métagénomique du microbiome intestinal des chatons a montré que le nombre de gènes uniques identifiés était environ 108 fois plus élevé que le nombre de cadres de lecture ouverts identifiés dans le génome du chat.10
Les phyla bactériens les plus abondants du microbiome intestinal sont les Firmicutes, les Bacteroïdetes et les Fusobacteria, suivis des Proteobacteria et des Actinobactéries.11
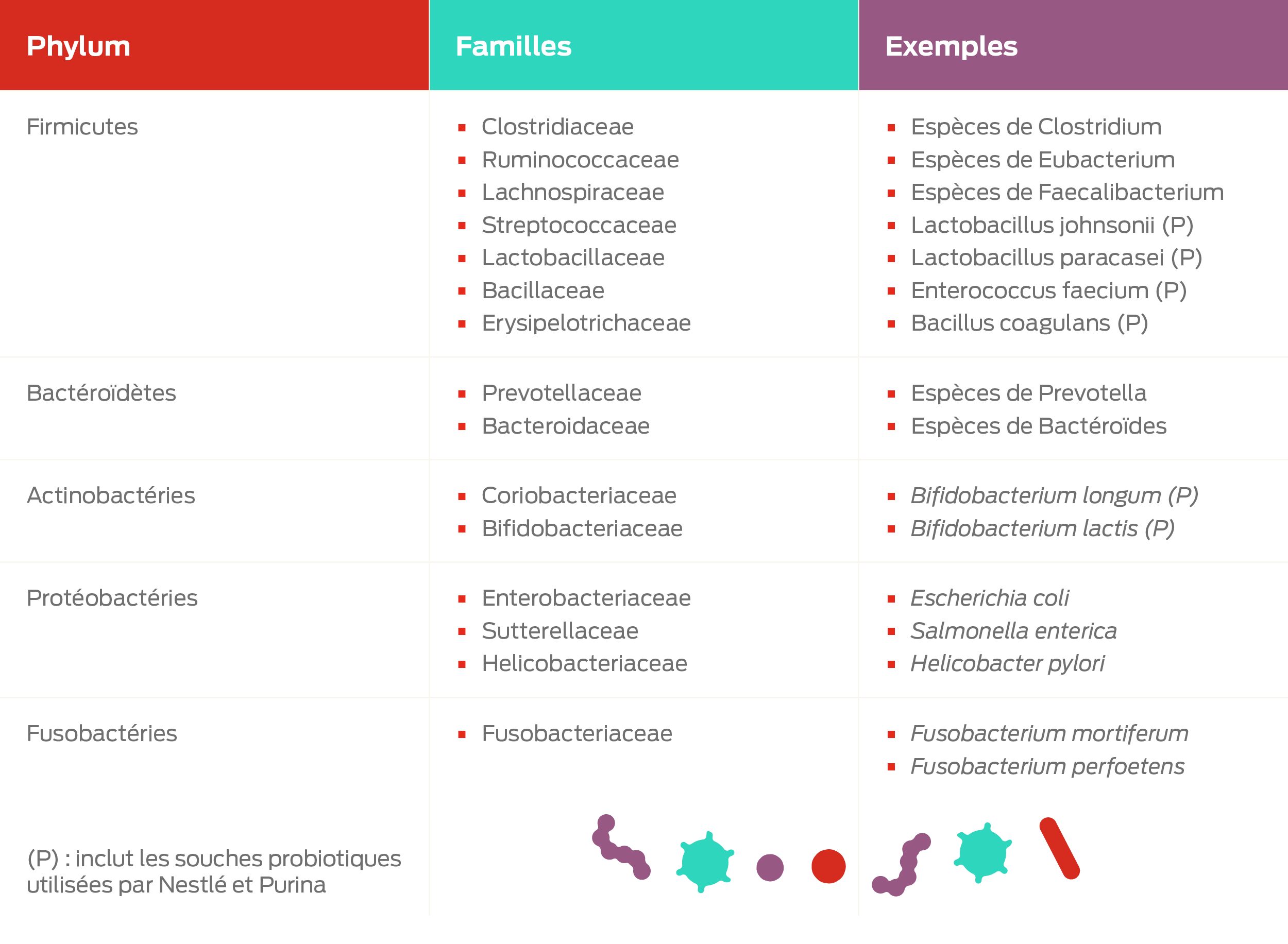
Le phylum Proteobacteria est le phylum le plus diversifié du microbiome intestinal et comprend un certain nombre d'agents pathogènes opportunistes connus, comme Escherichia coli, Klebsiella, Salmonella et Campylobacter, ainsi que des bactéries qui jouent un rôle vital dans l'homéostasie intestinale.12 Le microbiome fécal félin peut être plus diversifié que celui du chien.12
Explorer d'autres sections du Microbiome Forum

Interventions Portant sur le Microbiome pour Soigner les Pathologies des Animaux de Compagnie

Nestle et Purina, Leaders dans le Domaine du Microbiome
En savoir plus
- Barko, P.C., McMichael, M.A., Swanson, K.S., Williams, D.A. (2018). The gastrointestinal microbiome: a review. Journal of Veterinary Internal Medicine, 32, 9–25. doi: 10.1111/jvim.14875
- Marchesi, J. R. & Ravel, J. (2015). The vocabulary of microbiome research: a proposal. Microbiome, 3, 31. doi: 10.1186/s40168-015-0095-5
- Kim, J. Y., Whon, T. W., Lim, M. Y., Kim, Y. B., Kim, N., Kwo, M.-S.,…Na, Y.-D. (2020). The human gut archaeome: identification of diverse haloarchaea in Korean subjects. Microbiome, 8, 114. doi: 10.1186/s40168-020-00894-x
- Seth, E. C., & Taga, M. E. (2014). Nutrient cross-feeding in the microbial world. Frontiers in Microbiology, 5, 350. doi: 10.3389/fmicb.2014.00350
- Moreno, P. S., Wagner, J., Mansfield, C. S., Stevens, M., Gilkerson, J. R., & Kirkwood, C. D. (2017). Characterization of the canine faecal virome in healthy dogs and dogs with acute diarrhoea using shotgun metagenomics. PLoS ONE, 12(6), e0178433. doi: 10.1371/journal.pone.0178433
- Moreno, P. S., Wagner, J., Kirkwood, C. D., Gilkerson, J. R., & Mansfield, C. S. (2018). Characterization of the fecal virome in dogs with chronic enteropathy. Veterinary Microbiology, 221, 38–43. doi: 10.1016/j.vetmic.2018.05.020
- Zhang, W., Li, L., Deng, X., Kapusinszky, B., Pesavento, P. A., Delwart, E. (2014). Faecal virome of cats in an animal shelter. Journal of General Virology, 95, 2553–2564. doi: 10.1099/vir.0.069674-0
- Foster, M. L., Dowd, S. E., Stephenson, C., Steiner, J. M., & Suchodolski, J. S. (2013). Characterization of the fungal microbiome (mycobiome) in fecal samples from dogs. Veterinary Medicine International, 2013, 658373. doi: 10.1155/2013/658373
- Richards, P., Thornberry, N. A., & Pinto, S. (2021). The gut-brain axis: Identification of new therapeutic approaches for Type 2 diabetes, obesity, and related disorders. Molecular Metabolism, E pub ahead of print. doi: 10.1016/j.molmet.2021.101175
- Deusch, O., O'Flynn, C., Colyer, A., Morris, P., Allaway, D., Jones, P. G., & Swanson, K. S. (2014). Deep Illumina-based shotgun sequencing reveals dietary effects on the structure and function of the fecal microbiome of growing kittens. PLoS ONE, 9(7), e101021. doi: 10.1371/ journal.pone.0101021
- Suchodolski, J. S. (2011). The intestinal microbiota of dogs and cats: A bigger world than we thought. Veterinary Clinics of North America Small Animal Practice, 41, 261–272. doi: 10.1016/j.cvsm.2010.12.006
- Belas, A., Marques, C., & Pomba, C. (2020). The gut microbiome and antimicrobial resistance in companion animals. In Duarte, A. & Lopes da Costa, L. (Eds.), Advances in Animal Health, Medicine and Production (1st ed.), pp. 233–245. Springer International Publishing
