VILLI INTESTINALI CON BATTERI
Quali Elementi Compongono il Microbioma?
Il microbioma è la raccolta completa dei microrganismi (il microbiota), i loro geni e il loro microambiente (habitat) in un'area specifica. I batteri costituiscono circa il 98% del microbioma.1,2
I sottogruppi del microbioma comprendono il viroma (virus), il micobioma (funghi) e l'archeoma (archei; questi organismi somigliano ai batteri, ma sono un dominio distinto).3 Si tratta di un ambiente dinamico con interazioni complesse e metabolismi interconnessi.4
Il viroma comprende i virus che infettano i batteri (batteriofagi) all'interno del microbioma e le cellule dell'ospite nell'ambiente.5,6 I batteriofagi rappresentano la più ampia porzione del viroma.5,6 Ad oggi, la ricerca sul viroma è limitata,6,7 ma sono state identificate modificazioni del viroma fecale nei cani con enteropatia cronica o diarrea acuta.5,6 Analogamente, al momento risultano limitate anche le ricerche pubblicate sul micobioma degli animali domestici.8
Il numero di geni presenti nel microbioma intestinale supera di gran lunga quello dei geni dell'ospite. Nell'essere umano, si stima che il numero dei geni microbici sia oltre 100 volte più alto.9
L'analisi metagenomica del microbioma intestinale dei gattini ha dimostrato che il numero di geni unici identificati è circa 108 volte superiore al numero di cosiddette ORF (open reading frame) identificate nel genoma del gatto.10
I phyla batterici più abbondanti del microbioma intestinale sono Firmicutes, Bacteroides e Fusobacteria, seguiti da Proteobacteria e Actinobacteria.11
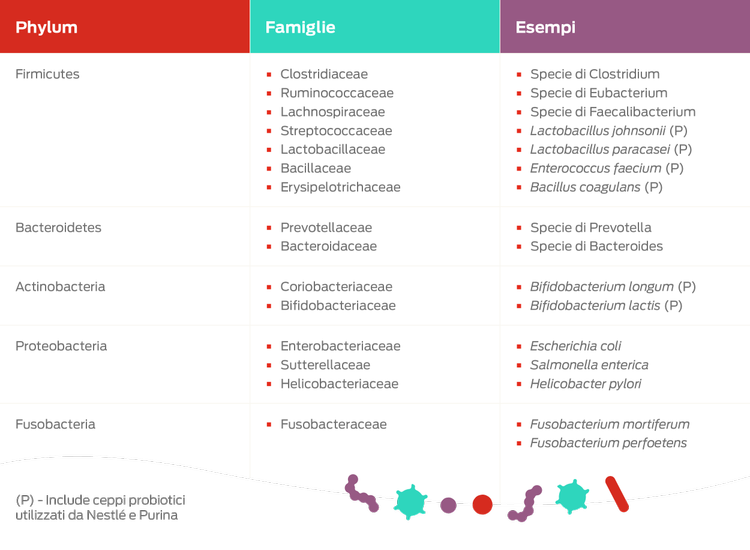
Il phylum Proteobacteria è il più eterogeneo del microbioma intestinale e comprende numerosi patogeni opportunistici noti, quali Escherichia coli, Klebsiella, Salmonella e Campylobacter, nonché batteri che svolgono un ruolo vitale nell'omeostasi intestinale.12 Il microbioma fecale del gatto potrebbe essere più composito di quello del cane.12
Scopri Altre Aree del Forum sul Microbioma

Interventi Incentrati sul Microbioma per le Condizioni di Salute Degli Animali Domestici
Ulteriori informazioni
- Barko, P.C., McMichael, M.A., Swanson, K.S., Williams, D.A. (2018). The gastrointestinal microbiome: a review. Journal of Veterinary Internal Medicine, 32, 9-25. doi: 10.1111/jvim.14875
- Marchesi, J. R. & Ravel, J. (2015). The vocabulary of microbiome research: a proposal. Microbiome, 3, 31. doi: 10.1186/s40168-015-0095-5
- Kim, J. Y., Whon, T. W., Lim, M. Y., Kim, Y. B., Kim, N., Kwo, M.-S.,…Na, Y.-D. (2020). The human gut archaeome: identification of diverse haloarchaea in Korean subjects. Microbiome, 8, 114. doi: 10.1186/s40168-020-00894-x
- Seth, E. C., & Taga, M. E. (2014). Nutrient cross-feeding in the microbial world. Frontiers in Microbiology, 5, 350. doi: 10.3389/fmicb.2014.00350
- Moreno, P. S., Wagner, J., Mansfield, C. S., Stevens, M., Gilkerson, J. R., & Kirkwood, C. D. (2017). Characterization of the canine faecal virome in healthy dogs and dogs with acute diarrhoea using shotgun metagenomics. PLoS ONE, 12(6), e0178433. doi: 10.1371/journal.pone.0178433
- Moreno, P. S., Wagner, J., Kirkwood, C. D., Gilkerson, J. R., & Mansfield, C. S. (2018). Characterization of the fecal virome in dogs with chronic enteropathy. Veterinary Microbiology, 221, 38-43. doi: 10.1016/j.vetmic.2018.05.020
- Zhang, W., Li, L., Deng, X., Kapusinszky, B., Pesavento, P. A., Delwart, E. (2014). Faecal virome of cats in an animal shelter. Journal of General Virology, 95, 2553-2564. doi: 10.1099/vir.0.069674-0
- Foster, M. L., Dowd, S. E., Stephenson, C., Steiner, J. M., & Suchodolski, J. S. (2013). Characterization of the fungal microbiome (mycobiome) in fecal samples from dogs. Veterinary Medicine International, 2013, 658373. doi: 10.1155/2013/658373
- Richards, P., Thornberry, N. A., & Pinto, S. (2021). The gut-brain axis: Identification of new therapeutic approaches for Type 2 diabetes, obesity, and related disorders. Molecular Metabolism, Pubblicazione in formato elettronico prima della stampa. doi: 10.1016/j.molmet.2021.101175
- Deusch, O., O’Flynn, C., Colyer, A., Morris, P., Allaway, D., Jones, P. G., & Swanson, K. S. (2014). Deep Illumina-based shotgun sequencing reveals dietary effects on the structure and function of the fecal microbiome of growing kittens. PLoS ONE, 9(7), e101021. doi: 10.1371/ journal.pone.0101021
- Suchodolski, J. S. (2011). The intestinal microbiota of dogs and cats: A bigger world than we thought. Veterinary Clinics of North America Small Animal Practice, 41, 261-272. doi: 10.1016/j.cvsm.2010.12.006
- Belas, A., Marques, C., & Pomba, C. (2020). The gut microbiome and antimicrobial resistance in companion animals. In Duarte, A. & Lopes da Costa, L. (Eds.), Advances in Animal Health, Medicine and Production (1st ed.), pp. 233–245. Springer International Publishing
